Осташ Богдан Омелянович
Посада: головний науковий співробітник кафедри генетики та біотехнології, професор кафедри генетики та біотехнології
Науковий ступінь: доктор біологічних наук
Вчене звання: доцент
Телефони (робочі): (032) 239-44-07, (032) 239-44-75
Електронна пошта: bohdan.ostash@lnu.edu.ua
Профіль у Google Scholar: scholar.google.com.ua
Профіль ORCID: orcid.org
Профіль у Scopus: www.scopus.com
Профіль у Web of Science (Publons): www.webofscience.com
Профіль у ResearchGate: www.researchgate.net
Наукові інтереси
Генетика вторинного метаболізму актинобактерій
Актинобактерії – одні із “найобдарованіших” хіміків Природи: вони синтезують величезну кількість хімічно і біологічно різноманітних природних сполук (див. приклади на рисунку внизу), відомих як вторинні метаболіти. Це сполуки, що не є життєво важливими для організму-продуцента, але, очевидно, надають йому селективну за певних умов існування. Антибіотики є, мабуть, найвідомішим і найціннішим класом вторинних метаболітів.
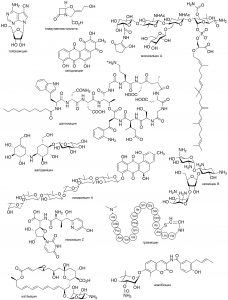
Приклади антибіотиків, що продукуються стрептоміцетами. Біосинтез деяких наведених сполук (ландоміцин, моеноміцин, амінокумаринові антибіотики, поліени) вивчається в НДЛ-42
У фокусі моїх досліджень – регуляція біосинтезу антибіотиків у роді Streptomyces. Зокрема, поточні наукові проекти зосереджено на вивченні усіх аспектів транскрипційної та посттранскрипційної регуляції експресії AdpA (відомого також як BldH) – плейотропного транскрипційного фактора, майстер-регулятора вторинного метаболізму та морфогенезу стрептоміцетів.
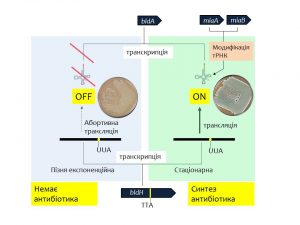
Концепція транскрипційно-трансляційного контролю синтезу біологічно активних речовин та морфогенезу в Streptomyces. Гени bldA й bldH складають центральну регуляторну пару – т.зв. “генний перемикач”. За відсутності зрілої тРНКLeuUAA (BldA) – до етапу пізньої експоненційної фази або за умов делеції гена bldA – UUA-вмісний транскрипт bldH не транслюється, або ж містранслюється з низькою частотою. Як результат, не відбувається перехід колонії стрептоміцета з фази субстратного міцелію у фазу росту повітряних гіфів. У цьому стані “генний перемикач” вимкнено (OFF). У стаціонарній фазі відбувається накопичення трансляційно компетентної (зрілої) тРНКLeuUAA. Посттранскрипційні модифікації тРНК (ПТМТ) – зокрема ті, що контролюють гени miaA й miaB – один із факторів появи зрілої тРНКLeuUAA. Остання, у свою чергу, забезпечує ефективну трансляцію мРНК bldH; BldH активує транскрипцію генів морфогенезу та вторинного метаболізму. У цьому стані “генний перемикач” увімкнено (ON)
Крім регуляції біосинтезу вторинних метаболітів, у фокусі досліджень перебувають гени і білки, що контролюють незвичні біохімічні реакції. Наприклад, нами показано, що приєднання низки хімічно відмінних донорів аміногрупи до термінального залишку уронової кислоти в фосфогліколіпідному антибіотику моеноміцині контролює один фермент, МоеН5 (див. рис. внизу), який репрезентує унікальний випадок еволюції глутамінових амідотрансфераз у каталізатор амідолігазного типу (Ostash et al. 2013).
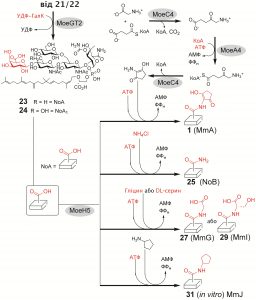
Амідотрансфераза МоеН5 переносить низку донорів аміногрупи на карбоксил галактуронової кислоти моеноміцину. Природні донорні субстрати – пентациклічний хромофор (А-кільце), іон амонію і гліцин. За умов in vitro МоеН5 здатний переносити інші групи
Еволюція кодонного складу
Кодувальні послідовності генів складаються з кодонів – трьох нуклеотидів поспіль, які визначають тип амінокислоти, що включатиметься у поліпептид під час трансляції. Багато амінокислот кодуються більше ніж одним кодоном (синоніми), і тому різні нуклеотидні послідовності можуть кодувати ідентичний поліпептид. Синонімічні кодони вживаються нерівномірно у різних видах, в межах одного генома і навіть гена – явище, відоме як переважне вживання кодонів (ПВК). ПВК – своєрідний шар додаткової генетичної інформації, що впливає, наприклад, на фолдинг, час початку і швидкість синтезу білка, а відтак має суттєві фенотипові прояви, зокрема й патологічні в людини (див. Plotkin and Kudla 2011). Нині немає задовільного пояснення глобальних причин і рушійних сил, що зумовили ПВК. Наявні експериментальні моделі (дріжджі, E. coli) – надто складні, і не дозволяють вивчення ПВК за допомогою повного видалення певного кодона (чи нокауту відповідного гена тРНК) з генома. Наші дослідження – внесок у розуміння ПВК на основі вивчення геномів з екстремальним зсувом у бік вживання GC-багатих кодонів. Як модель ми використовуємо Streptomyces albus J1074. Також значні зусилля докладаються до опрацювання нових методів візуалізації зсувів у вживанні та заміщенні кодонів – див. напр. Zaburannyi et al. 2009, а також cTools на http://biotools.online/
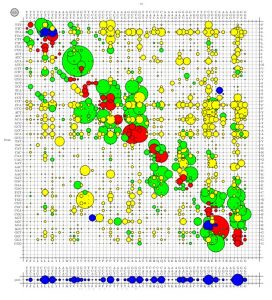
Bubble-plot графік, що відображає різні за частотою і типом заміщення кодонів у масиві гомологічних послідовностей. Графік – результат функціонування веб-застосунка X-Rate на сервері cTools (див. основний текст вище)
Курси
Публікації
Внизу подано вибрані наукові публікації за останні 5 років (2019-2023 рр); повний список публікацій – див. https://www.researchgate.net/profile/Bohdan_Ostash/contributions а також https://www.ncbi.nlm.nih.gov/pubmed/?term=ostash+b
Статті
- Dolya B, Hryhorieva O, Sorochynska K, Lopatniuk M, Ostash I, Tseduliak V-M, Sterndorff EB, Jørgensen TS, Gren T, Dacyuk Y, Weber T, Luzhetskyy A, Fedorenko V, Ostash B. Properties of multidrug-resistant mutants derived from heterologous expression chassis strain Streptomyces albidoflavus. Microorganisms. 2023; 11(5):1176. https://doi.org/10.3390/microorganisms11051176
- Koshla O, Vogt LM, Rydkin O, Sehin Y, Ostash I, Helm M, Ostash B. Landscape of post-transcriptional tRNA modifications in Streptomyces albidoflavus J1074 as portrayed by mass spectrometry and genomic data mining. J Bacteriol. 2023 Jan 26;205(1):e0029422. doi: 10.1128/jb.00294-22.
- Tseduliak VM, Dolia B, Ostash I, Lopatniuk M, Busche T, Ochi K, Kalinowski J, Luzhetskyy A, Fedorenko V, Ostash B. Mutations within gene XNR_2147 for TetR-like protein enhance lincomycin resistance and endogenous specialized metabolism of Streptomyces albus. J Appl Genet. 2023 Feb;64(1):185-195. doi: 10.1007/s13353-022-00738-4.
- Yushchuk O, Zhukrovska K, Ostash B, Fedorenko V, Marinelli F. Heterologous expression reveals ancient properties of Tei3 – a VanS ortholog from the teicoplanin producer Actinoplanes teichomyceticus. Int J Mol Sci. 2022 Dec 11;23(24):15713. doi: 10.3390/ijms232415713.
- Ostash B, Makitrynskyy R, Yushchuk O, Fedorenko V. Structural diversity, bioactivity, and biosynthesis of phosphoglycolipid family antibiotics: recent advances. BBA Adv. 2022 Nov 17;2:100065. doi: 10.1016/j.bbadva.2022.100065.
- Tsugita A, Uehara S, Matsui T, Yokoyama T, Ostash I, Deneka M, Yalamanchili S, Bennett CS, Tanaka Y, Ostash B. The carbohydrate tail of landomycin A is responsible for its interaction with the repressor protein LanK. FEBS J. 2022 Oct;289(19):6038-6057. doi: 10.1111/febs.16460.
- Shemediuk A, Dolia B, Ochi K, Fedorenko V, Ostash B. Properties of spontaneous rpsL mutant of Streptomyces albus KO-1297. Cytol Genet. 2022;56(1):31-36. doi: 10.3103/S009545272201011X.
- Melnyk S, Stepanyshyn A, Yushchuk O, Mandler M, Ostash I, Koshla O, Fedorenko V, Kahne D, Ostash B. Genetic approaches to improve clorobiocin production in Streptomyces roseochromogenes NRRL 3504. Appl Microbiol Biotechnol. 2022 Feb;106(4):1543-1556. doi: 10.1007/s00253-022-11814-4.
- Hrab P, Rückert C, Busche T, Ostash I, Kalinowski J, Fedorenko V, Yushchuk O, Ostash B. Complete genome sequence of Streptomyces cyanogenus S136, producer of anticancer angucycline landomycin A. 3 Biotech. 2021;11(6):282. doi: 10.1007/s13205-021-02834-4.
- Koshla O, Lopatniuk M, Borys O, Misaki Y, Kravets V, Ostash I, Shemediuk A, Ochi K, Luzhetskyy A, Fedorenko V, Ostash B. Genetically engineered rpsL merodiploidy impacts secondary metabolism and antibiotic resistance in Streptomyces. World J Microbiol Biotechnol. 2021 Mar 17;37(4):62. doi: 10.1007/s11274-021-03030-5.
- Yushchuk O, Ostash I, Mösker E, Vlasiuk I, Deneka M, Rückert C, Busche T, Fedorenko V, Kalinowski J, Süssmuth RD, Ostash B. Eliciting the silent lucensomycin biosynthetic pathway in Streptomyces cyanogenus S136 via manipulation of the global regulatory gene adpA. Sci Rep. 2021 Feb 10;11(1):3507. doi: 10.1038/s41598-021-82934-6.
- Silov S, Zaburannyi N, Anisimova M, Ostash B. The Use of the rare TTA codon in Streptomyces genes: significance of the codon context? Indian J Microbiol. 2021 Mar;61(1):24-30. doi: 10.1007/s12088-020-00902-6.
- Ostash B. Pleiotropic regulatory genes as a tool for Streptomyces strains bioprospecting and improvem Curr. Biotech. – 2021. – Vol. 10. P. 18–31. DOI: 10.2174/2211550110666210217105112
- Koshla O, Kravets V, Dacyuk Y, Ostash I, Süssmuth R, Ostash B. Genetic analysis of Streptomyces albus J1074 mia mutants suggests complex relationships between post-transcriptional tRNAXXA modifications and physiological traits. Folia Microbiol. 2020 Dec;65(6):1009-1015. doi: 10.1007/s12223-020-00811-7.
- Yushchuk O, Ostash B, Truman AW, Marinelli F, Fedorenko V. Teicoplanin biosynthesis: unraveling the interplay of structural, regulatory, and resistance genes. Appl Microbiol Biotechnol. 2020 Apr;104(8):3279-3291. doi: 10.1007/s00253-020-10436-y.
- Yushchuk O, Homoniuk V, Ostash B, Marinelli F, Fedorenko V. Genetic insights into the mechanism of teicoplanin self-resistance in Actinoplanes teichomyceticus. J Antibiot. 2020 Apr;73(4):255-259. doi: 10.1038/s41429-019-0274-9.
- Yushchuk O, Homoniuk V, Datsiuk Y, Ostash B, Marinelli F, Fedorenko V. Development of a gene expression system for the uncommon actinomycete Actinoplanes rectilineatus NRRL B-16090. J Appl Genet. 2020 Feb;61(1):141-149. doi: 10.1007/s13353-019-00534-7.
- Lopatniuk M, Myronovskyi M, Nottebrock A, Busche T, Kalinowski J, Ostash B, Fedorenko V, Luzhetskyy A. Effect of “ribosome engineering” on the transcription level and production of S. albus indigenous secondary metabolites. Appl Microbiol Biotechnol. 2019 Sep;103(17):7097-7110. doi: 10.1007/s00253-019-10005-y.
- Koshla O, Yushchuk O, Ostash I, Dacyuk Y, Myronovskyi M, Jäger G, Süssmuth RD, Luzhetskyy A, Byström A, Kirsebom LA, Ostash B. Gene miaA for post-transcriptional modification of tRNA(XXA) is important for morphological and metabolic differentiation in Streptomyces. Mol Microbiol. 2019 Jul;112(1):249-265. doi: 10.1111/mmi.14266.
- Koshla O, Rokytskyy I, Ostash I, Busche T, Kalinowski J, Mosker E, Sussmuth RD, Fedorenko V, Ostash B. Secondary metabolome and transcriptome of Streptomyces albus J1074 in liquid medium SG2. Cyt Genet. 2019; 53(1):1-7. doi: 10.3103/S0095452719010080.
- Yushchuk O, Horbal L, Ostash B, Marinelli F, Wohlleben W, Stegmann E, Fedorenko V. Regulation of teicoplanin biosynthesis: refining the roles of tei cluster-situated regulatory genes. Appl Microbiol Biotechnol. 2019 May;103(10):4089-4102. doi: 10.1007/s00253-019-09789-w.
- Yushchuk O, Kharel M, Ostash I, Ostash B. Landomycin biosynthesis and its regulation in Streptomyces. Appl Microbiol Biotechnol. 2019 Jan 11. doi: 10.1007/s00253-018-09601-1.
- Sehin Y, Koshla O, Dacyuk Y, Zhao R, Ross R, Myronovskyi M, Limbach PA, Luzhetskyy A, Walker S, Fedorenko V, Ostash B. Gene ssfg_01967 (miaB) for tRNA modification influences morphogenesis and moenomycin biosynthesis in Streptomyces ghanaensis Microbiology. 2019 Feb;165(2):233-245. doi: 10.1099/mic.0.000747.
Монографії
- Yushchuk O, Ostash B. Glycopeptide antibiotics: genetics, chemistry, and new screening approaches // In: “Natural products from actinomycetes”; Eds.: R.V. Rai, J.A. Bai– Springer Nature Singapore Pte Ltd, 2022. – P. 411–446. ISBN: 978-981-16-6132-7 (eBook); https://doi.org/10.1007/978-981-16-6132-7_16
- Ostash B., Anisimova M. Visualizing codon usage within and across genomes: concepts and tools //In: “Statistical modeling and machine learning principles for bioinformatics techniques, tools and applications”. Algorithms for Intelligent Systems; Eds.: K.G. Srinivasa, G.M. Giddesh, S.R. Manisekar – Springer Nature Singapore Pte Ltd, 2020. – P. 213–288. ISBN: 978-981-15-2444-8 (Print); https://doi.org/10.1007/978-981-15-2445-5_13
Патенти
- Патент на винахід №119472 Україна, МПК (2006.01)C12P 1/06 C12P 19/64 C12N 15/80 C12R 1/465 Спосіб активування продукції сполук з антибіотичною та протигрибною властивостями у Streptomyces cyanogenus S136 / Ющук О., Осташ Б.; заявник і власник Львівський національний ун-т ім. І. Франка. – № a201702642, заявл. 21 березня 2017 р., опубл. 25.06.2019, Бюл. № 12. https://library.ukrpatent.org/document?fund=1&id =259458&name=%D0%BE%D1%81%D1%82%D0%B0%D1%88+%D0%B1
- International application № WO/2008/021367 (PCT/US2007/01799), C07K 14/00 (2006.01), C12N 15/52 (2006.01). Moenomycin biosynthesis-related compositions and methods of use thereof / Inventors: B. Ostash, S. Walker. Filed and owned by Harvard University. – № WO/2008/021367, filed on 08.2007; published on 25.08.2015. Valid until 2031. https://patents.google.com/patent/US20100279980A1/en
Біографія
Народився в 1977р. В 1994р. вступив до Львівського державного університету ім. І. Я. Франка (тепер – Львівський національний університет імені Івана Франка (ЛНУ)). В 1999 р. закінчив ЛНУ і в цьому ж році вступив до аспірантури на кафедру генетики та біотехнології ЛНУ. Кандидатську дисертацію (спеціальність – 03.00.15) на тему “Генетичний контроль окремих етапів біосинтезу ландоміцину Е в Streptomyces globisporus 1912″ (науковий керівник – проф. Федоренко В.О.) захистив восени 2003 р. Докторську дисертацію (спеціальність – 03.00.22) на тему “Комбінаторний біосинтез нових антибіотиків у актиноміцетів” захистив восени 2012. З 2003 по 2004 р. – м.н.с. лабораторії генетики, селекції та генетичної інженерії продуцентів антибіотиків ЛНУ (НДЛ-42). З 2004 по 2007 рр. працював у відділі мікробіології та молекулярної генетики Гарвардської медичної школи (лабораторія проф. С’юзен Уокер). З 2007 р. – с.н.с. НДЛ-42, а з 2011 (за сумісництвом) – викладач кафедри генетики та біотехнології. Викладає спецкурси “Геноміка”, “Молекулярна філогенетика” та “Біоінформатика”. Галузі наукових інтересів – генетика бактерій, молекулярні механізми трансляційної регуляції метаболізму, біосинтез антибіотиків актинобактеріями. Перелік наукових публікацій і доступ до них – див. розділ Наука. Під керівництвом Осташа Б.О. захищено п’ять дисертацій на здобуття наукового ступеня кандидата біологічних наук (доктора філософії: Марія Рабик, 2012; Нестор Забуранний, 2013; Галина Мутенко 2017; Ігор Рокицький 2019, Оксана Кошла 2020). Наразі під керівництвом Осташа Б.О. працює два аспіранти.
Проекти
Керівництво
135/0009: “Drosophila melanogaster як платформа скринінгу нових противірусних сполук та вивчення клітинних механізмів захисту від вірусів”, 2023 р, Національний фонд досліджень України
БГ-21Ф: “Ген фосфорибозилізомерази, priA, як нове знаряддя метаболічної інженерії стрептоміцетів”, 2022 р, МОН України
БГ-80Ф: “Індукція мовчазних генів актинобактерій як метод виявлення нових біологічно активних сполук”, 2019 р, МОН України
Бг-41Нр: “Універсальний генетичний механізм контролю продукції біологічно активних речовин актиноміцетами”, 2016 р, МОН України
1/Бг4-18 “Генотипування мікробних ізолятів”, 2018 р., ПАТ “Галичфарм”
Ф80/2-2018 “Посттранскрипційні модифікації тРНК як регулятори первинного й вторинного метаболізму в актинобактерій”, 2017, Державний фонд фундаментальних досліджень України у рамках співпраці з Японським т-вом сприяння науки (JSPS)
М/18-2017+М/26-2018 “Плейотропні транскрипційні регулятори родини AdpA як знаряддя відкриття нових природних біоактивних сполук”, 2017 р., МОН України
Ф60/2-2015+Ф60/52-2016 “Рибосома як регуляторний елемент метаболізму: нове розуміння на основі підходів геномної інженерії”, 2015 р., Державний фонд фундаментальних досліджень України у рамках співпраці з Японським т-вом сприяння науки (JSPS)
М256-2013+М83-2014 “Ландоміцин А – платформа для розробки нових протиракових препаратів”, 2013 р., МОН України
Участь
R03TW009424 “Enabling biotechnologies to generate novel phosphoglycolipid antibiotics”, 2013-2015 рр., фінансувався фондом Фогарті при Національних інститутах здоров’я США. http://grantome.com/grant/NIH/R03-TW009424-03
INTAS – Ukraine 95-20: “Biotechnological improvement of novel potential anticancer antibiotic”. 1997-2000 рр., Німеччина.
БГ-46Ф: “Нові гени актинобактерій, що контролюють продукцію і стійкість до антибіотиків-інгібіторів синтезу пептидоглікану ”, 2017 р., МОН України.
Бл-01Ф: “Са2+-транспортувальні системи та регуляції клітинного дихання
екзокринних залоз у нормі і за дії стресорних чинників”. 2014 р., МОН України.
M/82-2016 “Інженерія вторинного метаболізму стрептоміцетів з використанням мутацій у гені рибосомного білка S12”, 2016 р., МОН України.
Бг-98Ф “Структурна і функціональна геноміка біосинтезу антибіотиків фосфогліколіпідної родини”, 2011 р., МОН України
Бг-01Ф, “Генетичні механізми стійкості актиноміцетів до моеноміцинів та роль рідкісних кодонів у їхньому біосинтезі”, 2009 р., МОН України
Бг-48П: “Біосинтез тіопептидів як спосіб утилізації відновлених сполук сірки: нові енергоощадні біотехнології для гірничої промисловості”, 2010 р., МОН України
Бг35-Ф “Гени актиноміцетів, що контролюють синтез агліконової частини ангуциклінових антибіотиків”, 2006 р., МОН України
Нагороди
1998 р. – стипендія Марії Ліон (Канада) для талановитих студентів з сільської місцевості
2002 р. – стипендія INTAS, що передбачала піврічне стажування в ун-ті Ов’єдо, Іспанія
2008 р. – премія Львівської облради для молодих науковців
2009 р. – стипендія Кабінету Міністрів України для талановитих молодих вчених
2009-2010 – стипендія від Броуд-інституту (США) у межах програми SPARC #2740563, що передбачала річне стажування у відділі мікробіології та молекулярної генетики Гарвардської медичної школи
2011 – індивідуальна стипендія Німецької служби академічних обмінів (DAAD) A/12/04489, що передбачала двомісячне стажування в Гельмгольц-інституті фармацевтичних досліджень (Саарбрюкен, ФРН)
2013 р. – іменна стипендія Верховної Ради України для найталановитіших молодих вчених
2014 р. – грамота ЛНУ ім. І. Франка за багаторічну сумлінну наукову діяльність
2017 р. – індивідуальна стипендія Німецької служби академічних обмінів (DAAD) N91655938, що передбачала двомісячне стажування в Центрі біотехнології ун-та м. Білефельд (ФРН)
2017 р. – премія Львівської міської ради талановитим вченим
2019 р. – стипендія Львівської системи дослідників
2020 р. – індивідуальна стипендія Німецької служби академічних обмінів (DAAD) N57507437, що передбачала двомісячне стажування в Майнцькому університеті (Німеччина).
Методичні матеріали
Великий практикум з генетики, генетичної інженерії та аналітичної біотехнології мікроорганізмів
Розклад
Сторінка розкладу викладачів не знайдена!